求む挑戦者!Nishika データ分析コンペティション「ソフトウェアの異常検知」
「あなたのデータサイエンスの力で企業の課題を解決する」
~Nishika Web サイトから~
こんにちは、井上 @michio_MWJ です。1222 年以来 800 年ぶりの「スーパー猫の日」はいかがでしたか? datetime を使って 22:22:22 の記念ツイートをされた方もいらっしゃいましたね(笑)私は既に寝落ちていました・・。
さて、今回は Nishika さんとの共同企画「ソフトウェアの異常検知」のご紹介とベンチマークコードの解説です。今日(2/25)からはじまるこのコンペ。ソフトウェアの安定的な稼働の為に異常を早く確実に検知すべく、ログデータをもとにしたシステムの健全性状態予測に挑戦します。
~2022/3/22 追記~
コード解説の YouTube Live を実施しました!
MATLAB ライセンス提供中!
どなたでも参加できますし使用ツールは問いません。が!! MATLAB の無料ライセンスも Toolbox もりもりで提供しておりますので、これを機に新しい Toolbox の味見がてらぜひ参加してみてください。(注:学生コンテストとの記載がありますがどなたでも利用可能です)
ライセンスに申し込む際のステップは 申し込みガイド にまめました。実際に利用いただけるまで申請から少し時間がかかりますのでまずは申請を済ませておくことをおススメいたします。
コンテスト概要
コンペの詳細・登録は「ソフトウェアの異常検知」から行ってください。
- 分析課題:ソフトウェアの異常検知
- 参加締切:2022年 4 月 26 日
- 賞金総額:25万円
MATLAB をメインに使って参加頂いた方には順位に応じて MATLAB 特別賞をお贈りします。
データは OS や WebLogic Server より観測される CPU やメモリの利用率など。それらを説明変数にしてソフトウェア(MATLAB じゃないよ)の異常を予測するというシンプルな設定。与えられた多くの時系列データをどう料理するかが問われます。

ベンチマーク解析
ここでは学習データの一部を使ってデータの読み込みから予測モデルの学習までを実施してみます。スクリプト自体はページ下部のボタンから DL できますし、使用するデータと一緒に MATLAB Drive でも公開しています。MathWorks アカウントと関連ライセンスがあれば MATLAB Online で実行してみることもできます。
今回、学習用データセットとして与えられている train.csv は 1.8 GB と多少大きいので、このコードでは一部を取り出した trainIphost17.csv を使用します。変数 host が “lphost17” のデータだけを抽出しています。読み込むデータを train.csv に置き換えてもそのまま機能するはずです。
使用する Toolbox
データの読み込み
datadir = “datafiles”; % ファイルの保存場所
filepath = fullfile(datadir,“trainlphost17.csv”);
ds = datastore(filepath)
170 変数が認識されていますね。問題なく読み込めそうです。preview で冒頭の 8 行だけ確認してみます。
preview(ds)
timestamp, host, Anomaly, process など170 個の変数が確認できますね。readall を使って一気に読み込んでみます。
matfilepath = fullfile(datadir,“trainData_lphost17.mat”);
if exist(matfilepath,“file”)
load(matfilepath,“trainData”);
else
trainData = readall(ds);
% mat ファイルからの読み込みの方が速いので保存しておく
save(matfilepath,“trainData”);
end
host と process がデータ元のラベルの様なのでこれらの種類を確認します。
unique(trainData.process)
unique(trainData.host)
このデータでは host は lphost17 のみ、そして process は wls1 と wls2 です。正常か異常のラベルが Anomaly という変数で、今回の目的変数ですね。ラベルの数を tabulate 関数で見ておきます。
tabulate(trainData.Anomaly)
異常データ(Anomaly = 1)がかなり少ないことが確認できます。データ元は host/process で特定できますが、2変数だと不便なのでデータ元を表す変数 origin を作っておきます。それぞれカテゴリ変数に変換しておきます。
trainData.origin = categorical(string(trainData.host) + string(trainData.process));
trainData.host = categorical(trainData.host);
trainData.process = categorical(trainData.process);
時系列の確認
ここでは host/process 毎にデータ、特に時刻データをチェックします。パッと見たところ時刻データはきれいに並んでいるようですが、timestamp の最大、最小、時間間隔を念のため確認します。
% グループ要約の計算
Gstats = groupsummary(trainData,[“host”,“process”],{“max”,“min”,…
@(x) {unique(diff(x))}},“timestamp”)
データ数は wls1/wls2 ともに大体同じ。計測開始・終了時刻も同じとなっています。時間ステップ幅を確認するために関数ハンドルを使って @(x) {unique(diff(x))} という処理を加えています。process = wls1 のデータを見ると
Gstats.fun1_timestamp{1}
と 1 分から 3 時間までばらつきますね。このサンプルコードではこの点について特に処置は行わずこのまま進めます。データの前処理については データの前処理:データ クリーニング、平滑化、グループ化で紹介している内容が役立つかもしれません。
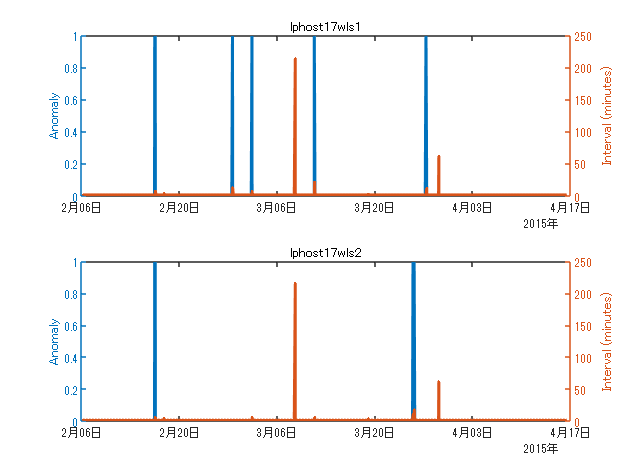
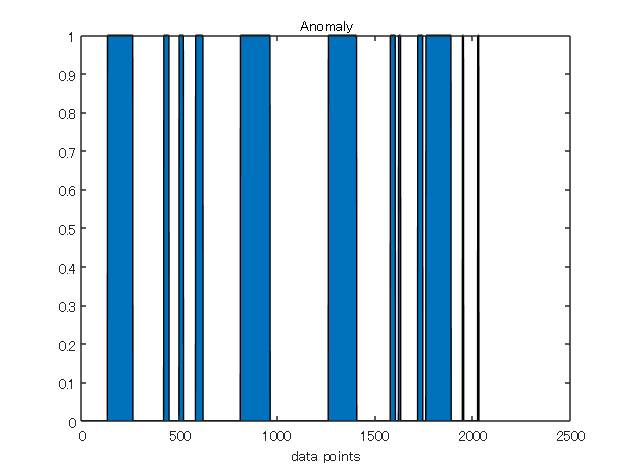
異常の発生状況プロット
学習データ内の異常発生状況を確認します。
uniqueOri = unique(trainData.origin);
figure(1)
tiledlayout(“flow”);
for ii=1:length(uniqueOri)
idx = trainData.origin == uniqueOri(ii);
tmp = trainData(idx,:);
nexttile
yyaxis left
plot(tmp.timestamp,tmp.Anomaly,LineWidth=2)
ylabel(“Anomaly”)
hold on
difftimestep = [0; diff(tmp.timestamp)];
yyaxis right
plot(tmp.timestamp, minutes(difftimestep),LineWidth=2)
ylabel(“Interval (minutes)”)
hold off
title(uniqueOri(ii))
end
wls1 では 5 回、wls2 では 2 回異常が発生しています。時間ステップの揺らぎ合わせてプロットしていますが、異常発生と同時に起こっている場合もあれば関係なく発生している箇所もあります。
説明変数の絞り込み
説明変数が約 170 個と多いので、サクッと計算させるために使用する変数を絞り込みます。絞り込む方法は多数考えられますがこちらのヘルプページ(特徴選択の紹介)も参考になるかもしれません。ここでは Anomaly と相関が高いもの、そして相互に相関が高いものは片方だけを残すことにします。timestamp, host, process 以外の数値データを対象とします。
S = vartype(“numeric”);
features = trainData(:,S); % 数値データだけ取り出す
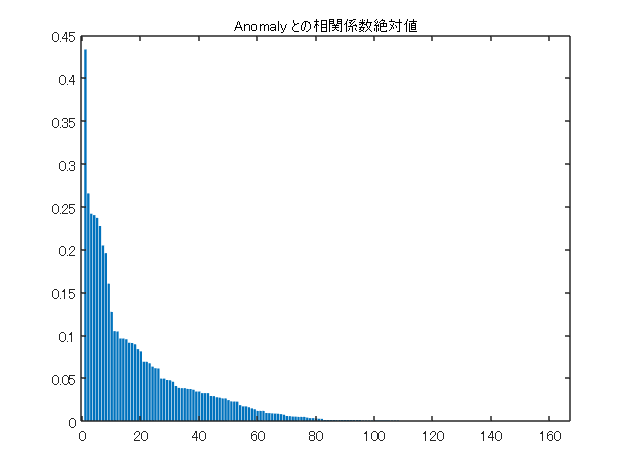
corrcoef 関数で相互相関を計算して、Anomaly との相関係数の絶対値(1行目)を昇順に並べます。
R = corrcoef(features.Variables);
[Rsort,idx] = sort(abs(R(1,:)),‘descend’,‘MissingPlacement’,‘last’);
figure
bar(Rsort(2:end))
title(“Anomaly との相関係数絶対値”)
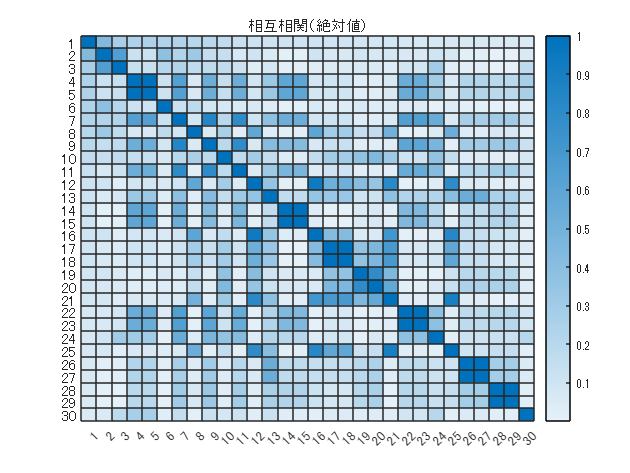
次に Anomaly との相関係数(絶対値)が上位 30 の変数に絞って、相互相関を見てみます。
features30 = features(:,idx(1:30));
figure
R = corrcoef(features30.Variables);
heatmap(abs(R))
title(“相互相関(絶対値)”)
説明変数間でも相関係数が高いものがあります。特に高い(0.9 以上)ものは同じ情報を持っているとみなして片方だけ使用することで更に変数を絞り込みます。
absR = abs(R);
tmp = triu(absR>0.9,1); % 行列の上三角部分
[row,col] = ind2sub(size(absR),find(tmp)); % 0.9 以上の位置
idxDiag = row == col;
row = row(~idxDiag);
col = col(~idxDiag); % 0.9 未満のものだけ残す
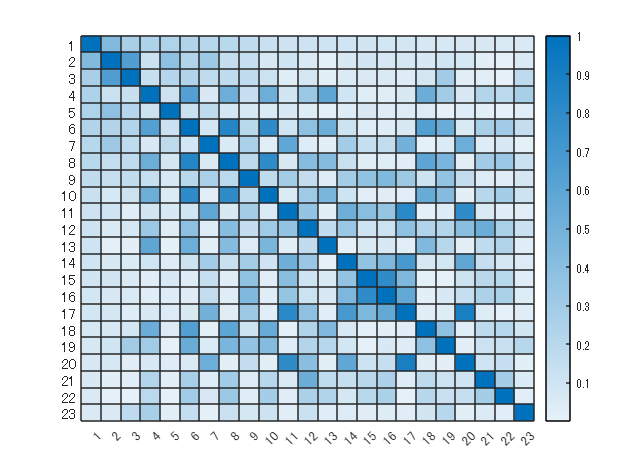
figure
features2use = features30;
features2use(:,col) = [];
R = corrcoef(features2use.Variables);
figure
heatmap(abs(R))
Anomaly 含めて 23 個残りました。変数の名前(Anomaly との相関(絶対値)が高い順)を確認しておきます。
varnames = features2use.Properties.VariableNames’
予測モデルの学習
次はこの変数で予測モデルを作ってみます。
学習に使用するデータ区間の切り出し
上で見た通り正常(Anomaly = 0)のデータが多いため、正常データのアンダーサンプリングの為以下の操作を行います。
- 異常発生箇所を確認し異常区間の長さ(データ点数)を確認
- 異常中(Anomaly = 1) の前後に異常区間の長さ分のデータを加えた区間を抽出
dataSeg = []; % データ確保用変数
dataOri = []; % データ元
dataAll = []; % データ確保用変数(単純に繋げたもの)
uniqueOri = unique(trainData.origin)
% 注意:スタートとエンドは正常であることが前提(lphost7 のデータはOK)
for ii=1:length(uniqueOri)
idx = trainData.origin == uniqueOri(ii);
tmp = trainData(idx,varnames);
% 以下を探します。
% 0から1に変化する箇所: anomaly starts
% 1から0に変化する箇所: anomaly ends
isAnomaly1 = tmp.Anomaly;
isAnomaly2 = [0; isAnomaly1(1:end-1)];
isChange = xor(isAnomaly1, isAnomaly2); % 変化点検知
posFalls = find(isChange & isAnomaly2); % 1 -> 0 (end)
posRises = find(isChange & ~isAnomaly2); % 0 -> 1 (start)
for jj=1:length(posRises)
duration = 1*(posFalls(jj) – posRises(jj));
if posRises(jj) – duration > 0
idxStart = posRises(jj) – duration;
else
idxStart = 1;
end
if posFalls(jj) + duration < height(tmp)
idxEnd = posFalls(jj) + duration;
else
idxEnd = height(tmp);
end
dataSeg = [dataSeg; {tmp(idxStart:idxEnd,:)}];
dataAll = [dataAll; tmp(idxStart:idxEnd,:)];
dataOri = [dataOri; uniqueOri(ii)];
end
end
tabulate(dataAll.Anomaly)
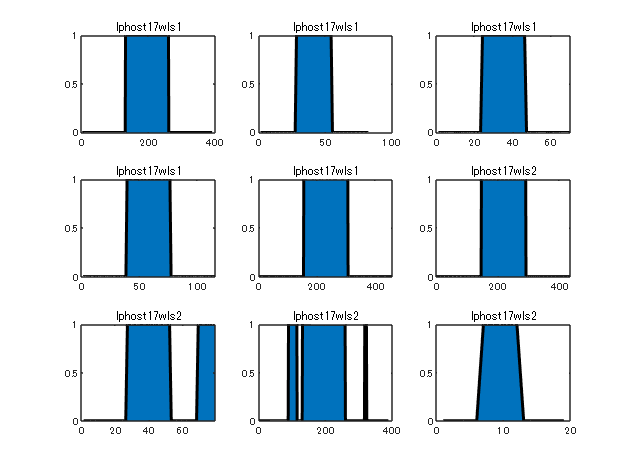
正常 (0) と異常 (1) のデータ比が 2:1 程度です。
切り出し部分のプロット
まず単純に繋げてプロット。
figure
area(dataAll.Anomaly)
title(“Anomaly”)
xlabel(“data points”)
異常箇所毎に分けてプロット。
figure
tiledlayout(‘flow’);
for ii=1:length(dataSeg)
nexttile
tmp = dataSeg{ii};
area(tmp.Anomaly,‘LineWidth’,2)
title(dataOri(ii));
end
hold off
予測モデル学習 LSTM
まずは時系列データとしてではなく、単純に各観測値を独立したものとみて予測モデルを作ってみるのも手です。このスクリプトでは省略しますがこちらのページ(モデルの作成と評価:特徴選択、特徴量エンジニアリング、モデル選択、ハイパーパラメーターの最適化)が参考になるかと思います。
ここでは LSTM を使って予測モデルを作ってみます。時系列データの分類問題として深層学習を使用した sequence-to-sequence 分類を参考します。時系列、シーケンス、およびテキストを使用した深層学習のページも時系列データの取り扱いに関する情報元として参考になるかもしれません。
データの分割
上で作った各異常区間のデータ(dataSeg)を使用しますが、まずは学習用とテスト用に分割します。ここではデータ元(lphost17wls1 or lphost17wls2)で層化させて、学習データと検証データを分割します。
rng(0) % 同じ結果が再現するように乱数シートを固定
cv = cvpartition(dataOri,‘Holdout’,0.4);
学習データ(6セット)
trainSeg = dataSeg(cv.training)
検証データ(3セット)
testSeg = dataSeg(cv.test)
LSTM の学習データとして使用するために、各時系列セグメントとラベルデータをセル配列として成形します。generateDataset4LSTM 関数はスクリプト下部にローカル関数として定義しています。
[xTrain,yTrain] = generateDataset4LSTM(trainSeg); % 学習データ
[xTest,yTest] = generateDataset4LSTM(testSeg); % テストデータ
こんな形です。セルの中身は 22 個の特徴量が 22 x N の形で並んでいることに注意。
xTrain
ネットワーク構築
試しに隠れユニット数 100 の LSTM 層を 2 層としてみます。この辺は最適化する余地はたくさんありそうです。実験マネージャーアプリが役に立つかもしれません。
numFeatures = 22; % 特徴量変数の数
numHiddenUnits = 100; % 隠れユニット数
numClasses = 2; % 予測クラス数(異常・正常)
layers = [ …
sequenceInputLayer(numFeatures)
lstmLayer(numHiddenUnits,‘OutputMode’,‘sequence’)
lstmLayer(numHiddenUnits,‘OutputMode’,‘sequence’)
fullyConnectedLayer(numClasses)
softmaxLayer
classificationLayer(‘Classes’,{‘0’,‘1’},‘ClassWeights’,[1,2])]
分類層の設定で重み付きクロスエントロピー損失関数を使用しています。詳細はこちらの classificationLayer のヘルプページ(英語)を確認してください。
学習オプション設定
options = trainingOptions(‘adam’, …
‘MaxEpochs’,10, …
‘MiniBatchSize’,1, …
‘ValidationData’, {xTest,yTest},…
‘Plots’,‘training-progress’);
学習
rng(0) % 同じ結果が再現するように乱数シートを固定
net = trainNetwork(xTrain,yTrain,layers,options);
保存しておきます。
save(‘lstmnet_lphost17_LSTM.mat’,‘net’,‘varnames’);
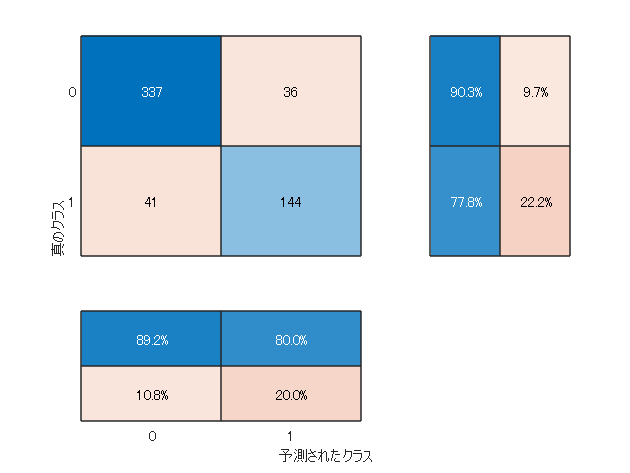
検証データへの予測精度を再確認
[yPred, scores] = classify(net,xTest);
figure
混同行列を描きます(異常区間ごとにセル配列になっているのですべて結合しています)
confusionchart([yTest{:}], [yPred{:}],…
‘ColumnSummary’,‘column-normalized’,…
‘RowSummary’,‘row-normalized’);
まあまぁです。
評価指標 PRC (Prediction-Recall-Curve)
今回の評価指標である PRC (Prediction-Recall-Curve) を描いてみます。
trueLabel = [yTest{:}];
labelScores = [scores{:}];
anomalyScores = labelScores(2,:); % labels(2) = 1 (異常) の score
N = 100; % 閾値の段階数
recalls = zeros(N,1);
precisions = zeros(N,1);
thresholds = linspace(0,1,N);
labels = net.Layers(end).Classes;
% 閾値を変えて recall/recision を計算
for ii=1:N
th = thresholds(ii);
[recalls(ii),precisions(ii)] = getRecallPrecisionCurve(th,anomalyScores,trueLabel,labels);
end
figure
plot(recalls, precisions)
提出用の予測作成
datadir フォルダ内に test.csv がある想定です。
load(‘lstmnet_lphost17_LSTM.mat’,‘net’,‘varnames’); % モデル読み込み
テストデータ読み込み
test.csv ファイルは コンペページ(ソフトウェアの異常検知)から参加登録後に入手して datadir に保存していることが前提です。
matfilepath = fullfile(datadir,“test.mat”);
if exist(matfilepath,“file”)
load(matfilepath,“testData”);
else
filepath = fullfile(datadir,‘test.csv’);
testData = readtable(filepath);
% mat ファイルからの読み込みの方が速いので保存しておく
save(matfilepath,“testData”);
end
学習に使用した変数 varnames と id/host/process のみを取り出します。
testData = testData(:,[{‘id’;‘host’;‘process’};varnames(2:end)]);% varname(1) は Anomaly
head(testData)
ここは思い切って、host/process もまとまったうえで、時系列順に id が並んでいるという想定で進めます・・。host/process 別に識別できるようデータ元(origin)変数を作っておきます。
testData.origin = categorical(string(testData.host) + string(testData.process));
予測
host/process 毎に切り分けて予測処理を実行します。
testOri = unique(testData.origin);
testData.Anomaly = zeros(height(testData),1);
for ii=1:length(testOri)
idx = testData.origin == testOri(ii);
tmpData = testData{idx,varnames(2:end)};
tmpData = zscore(tmpData); % 変数を正規化(方法は検討の余地あり)
[~,tmpScores] = classify(net,tmpData’);
testData.Anomaly(idx,:) = tmpScores(2,:)’; % labels(2) の score
end
提出ファイル作成
submission.csv に出力する値は Anomaly = 1 であるスコアなので、上の scores の2列目です。
output = testData(:,[“id”, “Anomaly”]);
head(output)
writetable(output,“submission.csv”);
まとめ
無事に最後の提出用ファイル作成までたどり着きました。今回はサクサク進めるように一部のデータだけを使ってみましたが、ぜひ本番の train.csv でも試してみてください。
もう少しデータを詳しく調査してもっとよい変数を探してみたり、新たな特徴量を作ってみたり、モデルに凝ってみたり、といろいろ試したいことが出てきますが、ここから先はコンペに参加する方のお楽しみということで私からの紹介はここまでとします。
いずれにしても一部の変数(or 一部のデータ)から始めてみて、まずデータの把握と課題の理解が第一歩かと思います。応募締め切りは 4 月 26 日。入賞目指して頑張ってください!
コンペへの参加登録はこちらから:ソフトウェアの異常検知
MATLAB ライセンス
冒頭でも触れましたが MATLAB を使って参加される方にはライセンスも提供しています。上でも触れた機械学習を行う Statistics and Machine Learning Toolbox™、そしてディープラーニングをカバーする Deep Learning Toolbox™ を含む 18 個の製品が利用可能です。是非ご活用ください。

関連リンク集
- Nishika ソフトウェアの異常検知
- MATLAB ライセンス申し込みページ と 申し込みガイド
- ベンチマークスクリプト(MATLAB Drive)
- ヘルプページ:データの前処理:データ クリーニング、平滑化、グループ化
- ヘルプページ:特徴選択の紹介
- ヘルプページ:モデルの作成と評価:特徴選択、特徴量エンジニアリング、モデル選択、ハイパーパラメーターの最適化
- ヘルプページ:深層学習を使用した sequence-to-sequence 分類
- ヘルプページ:時系列、シーケンス、およびテキストを使用した深層学習
ヘルパー関数
閾値を変えて recall と precision を計算する関数
function [recall,precision] = getRecallPrecisionCurve(threshold,anomalyScores,trueLabel,labels)
prediction = repelem(labels(1), length(trueLabel),1);
prediction(anomalyScores > threshold,:) = labels(2);
results = confusionmat(trueLabel,prediction);
TN = results(1,1);
TP = results(2,2);
FN = results(2,1);
FP = results(1,2);
precision = TP/(TP+FP);
recall = TP/(TP+FN);
end
LSTM を学習する為にデータを成形する関数
function [x,y] = generateDataset4LSTM(dataSeg)
x = [];
y = [];
for ii=1:length(dataSeg)
tmpT = dataSeg{ii};
% Anomaly 以外の変数を正規化(方法は検討の余地あり)
tmpT{:,2:end} = zscore(tmpT{:,2:end});
tmpX = tmpT{:,2:end}’;
tmpY = tmpT.Anomaly(:,:)’;
x = [x; {tmpX}];
y = [y; {categorical(tmpY)}];
end
end
- Category:
- Community,
- コンペティション





Comments
To leave a comment, please click here to sign in to your MathWorks Account or create a new one.